AI リサーチャーの松野です。 教師ラベルがない状況を想定して外れ値検出を試みる機会があったので、試したことをご紹介したいと思います。
対象としたサンプルデータについて詳細は述べませんが、十数次元のテーブルデータで、とあるラベル付けがされています。 このラベルによって抽出される2つのグループ A, B についてそれぞれ同一の外れ値検出を試みます。 データ件数は、グループAは約1万件、グループBは約4千件でした。
Isolation forest を用いた外れ値検出
まずは素直に、scikit-learn に実装されている isolation forest を適用してみます。 今回は正解データがあるので、外れ値検出の性能を評価することができました。
グループA
precision recall f1-score
False 0.9991 0.9373 0.9672
True 0.1225 0.9149 0.2161
accuracy 0.9371
macro avg 0.5608 0.9261 0.5917
weighted avg 0.9908 0.9371 0.9601
グループB
precision recall f1-score
False 0.9814 0.9084 0.9435
True 0.1117 0.4000 0.1746
accuracy 0.8942
macro avg 0.5465 0.6542 0.5590
weighted avg 0.9570 0.8942 0.9220
True が外れ値です。
外れ値はどちらのグループにおいても全体の数%しかないため、外れ値でないクラスの再現率 (recall) が高いが外れ値の適合率 (precision) が低くなっています。
外れ値を見逃すことは避けたいので、適合率が低くても外れ値の再現率が高ければよいことにします。
グループBの再現率が 40% と低いので、改良を検討します。
次元削減による可視化
データの性質を理解するため、t-SNE によって次元削減をおこなって可視化してみます。 左側が正解ラベル、右側が外れ値検出の結果です。
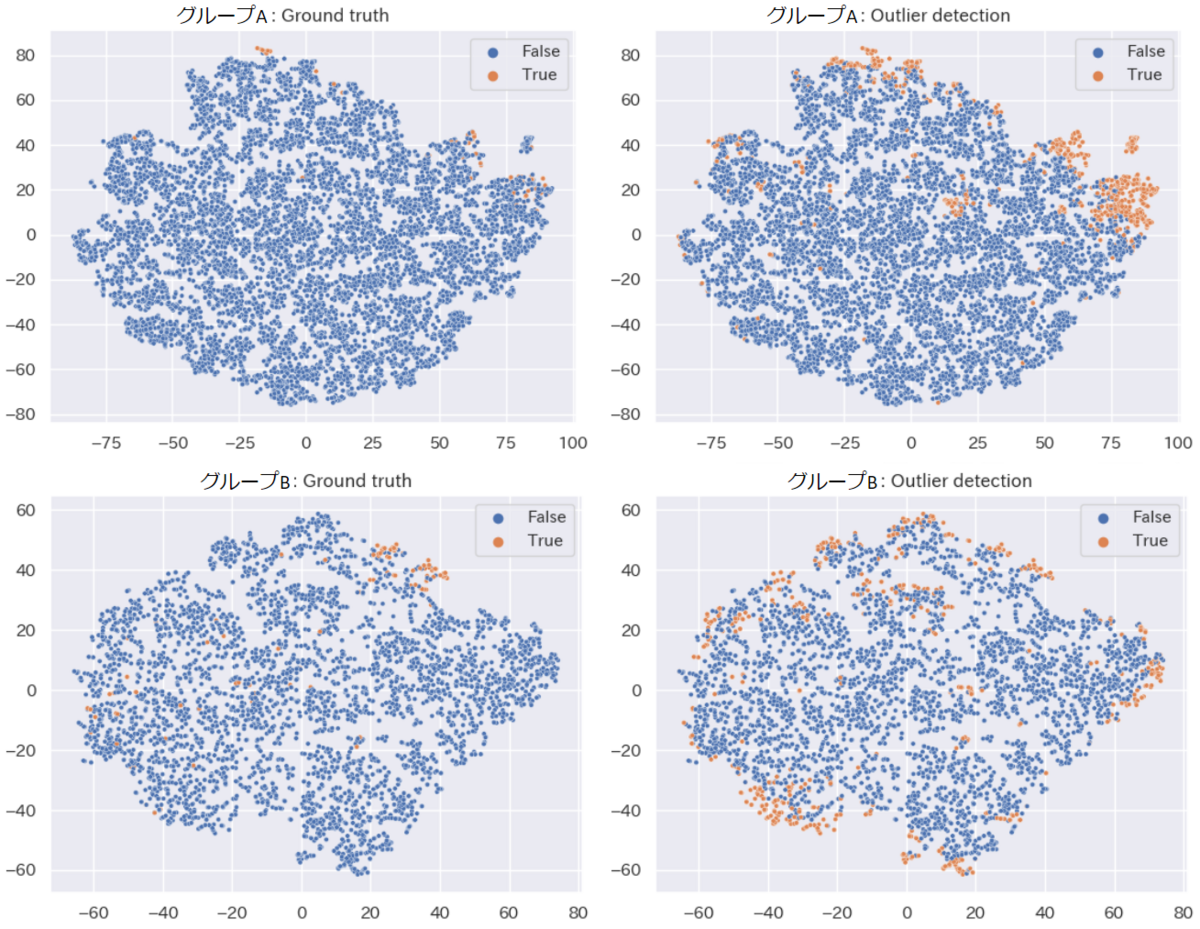
埋め込み空間において、外縁部に外れ値が比較的多く存在しているのがわかります。 そこで、次元削減後に外れ値検出を行うことで精度向上できないか検討してみました。
UMAP による次元削減の活用
次元削減の手法として有名なものに PCA, t-SNE, UMAP がありますが、以下の理由で UMAP を選択しました。
- 非線形な次元削減(PCA は線形)
- モデルを保存しておき、新しい入力データに対して同一の次元削減を実行できる(t-SNE は、あとから来たデータに対して同一の次元削減を実行できない)
UMAP のドキュメントを確認すると、外れ値検出と組み合わせるチュートリアルが紹介されています。
このチュートリアルでは MNIST データセットを対象に、Local Outlier Factor (LOF) 法によっておこなう外れ値検出が UMAP によって改善することや、その際に UMAP の set_op_mix_ratio パラメータをデフォルトから変更するのがよいことが紹介されています。
 図:UMAP + LOF によって MNIST データセットから検出された外れ値の例。
図:UMAP + LOF によって MNIST データセットから検出された外れ値の例。
同様の試みを今回のケースに対して行ってみました。 最初に紹介したモデルとアンサンブルするようにしています。 データ読込処理は省略してあります。
from sklearn.ensemble import IsolationForest from umap import UMAP # Outlier detection for original data iforest_high_dim = IsolationForest() iforest_high_dim.fit(X) inlier_score_original = iforest_high_dim.score_samples(X) + 1 # Outlier detection after dimensional reduction umap = UMAP(set_op_mix_ratio=0.25) X_umap = umap.fit_transform(X) iforest_low_dim = IsolationForest() iforest_low_dim.fit(X_umap) inlier_score_low_dim = iforest_low_dim.score_samples(X_umap) + 1 inlier_score = (inlier_score_original + inlier_score_low_dim) / 2 is_outlier = inlier_score < 0.5
結果は以下のようになりました。
グループA
precision recall f1-score
False 0.9998 0.8782 0.9351
True 0.0714 0.9787 0.1330
accuracy 0.8792
macro avg 0.5356 0.9285 0.5341
weighted avg 0.9910 0.8792 0.9275
グループB
precision recall f1-score
False 0.9890 0.8797 0.9311
True 0.1364 0.6609 0.2262
accuracy 0.8735
macro avg 0.5627 0.7703 0.5787
weighted avg 0.9652 0.8735 0.9114
グループAの適合率が犠牲になったものの、両グループの再現率、および、グループBの適合率を改善することができました。 しかし依然としてグループBの再現率は約 66% とあまり高くありません。 このグループに対する外れ値検出のむずかしさがあるようです。
進展があればまたの機会にご紹介したいと思います。
www.slideshare.net